三磷酸腺苷合酶
三磷酸腺苷合酶或ATP合酶,三磷酸腺苷酶(ATPase)的一种,在这里并特指F类的FoF1ATP合酶(F Type FoF1 ATP Synthase)。它利用呼吸链产生的质子的电化学势能,通过改变蛋白质的结构来进行三磷酸腺苷(ATP)的合成。ATP是大多数生物体中细胞最常用的“能量通货”。 它由二磷酸腺苷(ADP)和无机磷酸盐(Pi)形成。 ATP合酶催化的总体反应为:
- ADP + Pi + H+out ⇌ ATP + H2O + H+in
| ATP合酶 | |||||||
|---|---|---|---|---|---|---|---|
 | |||||||
| 通过X射线晶体学测定ATP合酶的分子模型 | |||||||
| |||||||
| 识别码 | |||||||
| EC编号 | 3.6.3.14 | ||||||
| CAS号 | 9000-83-3 | ||||||
| 数据库 | |||||||
| IntEnz | IntEnz浏览 | ||||||
| BRENDA | BRENDA入口 | ||||||
| ExPASy | NiceZyme浏览 | ||||||
| KEGG | KEGG入口 | ||||||
| MetaCyc | 代谢路径 | ||||||
| PRIAM | 概述 | ||||||
| PDB | RCSB PDB PDBj PDBe PDBsum | ||||||
| 基因本体 | AmiGO / EGO | ||||||
| |||||||
ATP合酶由两个主要的亚基Fo和F1组成,它们具有允许ATP产生的旋转运动机制[1][2]。
位置
在真核细胞中,ATP合酶存在于线粒体的内膜,Fo亚单位存在于膜内,F1
结构与功能
当前,原核生物的F型ATP酶的结构已经比较清楚了:
- F1单元 – α(3个)、β(3个)、γ(1个)、δ(1个)、ε(1个)
- Fo单元 – a(1个)、b(2个)、c(9-12个)
真核生物的F型ATP酶F1单元的种类的数量与原核生物相同、Fo单元的结构与原核生物相似,但是亚单位的数量不是很明瞭。
F1单元
ATP合酶的F1部分是亲水性的,并且负责水解ATP。 子单元和创建一个具有6个结合位点的hexameter。 其中三个是催化无活性的,并且它们结合成ADP。
| 子单元 | 人类基因 |
|---|---|
| alpha | ATP5A1, ATPAF2 |
| beta | ATP5B, ATPAF1, C16orf7 |
| gamma | ATP5C1 |
| delta | ATP5D |
| epsilon | ATP5E |
FO单元

FO是具有八个子单元的和跨膜环的水不溶性蛋白质。
| 子单元 | 人类基因 |
|---|---|
| a | ATP6 |
| b | ATP5F1 |
| c | ATP5G1, ATP5G2, ATP5G3 |
ATP合酶的反应
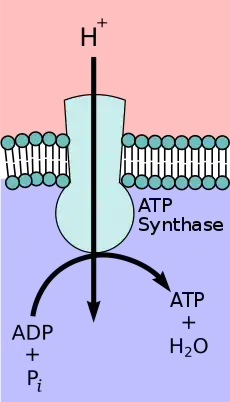
F1 单元催化以下ATP合成反应。
F1单元催化的反映是可逆的,在进行离子运输时,进行ATP水解反应。
Fo单元形成离子通道,质子可以从中通过:
- H+in H+out
当质子利用电化学势能通过Fo单元时,可以带动和其连接的轴(γ亚单位),改变F1单元的结构,进而调节F1单元与ATP和ADP:Pi的结合能(Binding Energy),降低ATP生成的活化能,达到ATP合成的目的:
- ADP + Pi + 3 H+out → ATP + 3 H+in
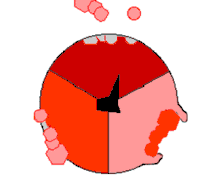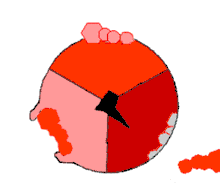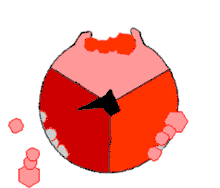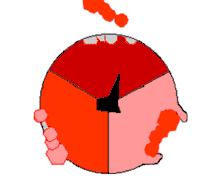
结合模型
在ATP酶的酶学模型中,验证其γ轴是否旋转占有重要地位,1997年,英国自然杂志(vol. 386, pp. 299–302)刊了日本科学家题为"Direct observation of the rotation of F1-ATPase"的文章,报道了ATP合成酵素F1单元可以通过水解ATP造成γ亚单位(轴)的旋转,并进行了单分子观察和录像、该論文证明了保罗·博耶的「ATP合酶的旋转理论」。同年保罗·博耶,约翰·沃克和延斯·克里斯蒂安·斯科因ATP合酶的研究获得诺贝尔化学奖。
參考資料
- Okuno D, Iino R, Noji H. . Journal of Biochemistry. April 2011, 149 (6): 655–64. PMID 21524994. doi:10.1093/jb/mvr049.
- Junge W, Nelson N. . Annual Review of Biochemistry. June 2015, 84: 631–57. PMID 25839341. doi:10.1146/annurev-biochem-060614-034124.
- PDB 1VZS; Carbajo RJ, Silvester JA, Runswick MJ, Walker JE, Neuhaus D. . Journal of Molecular Biology. 2004, 342 (2): 593–603. PMID 15327958. doi:10.1016/j.jmb.2004.07.013.
